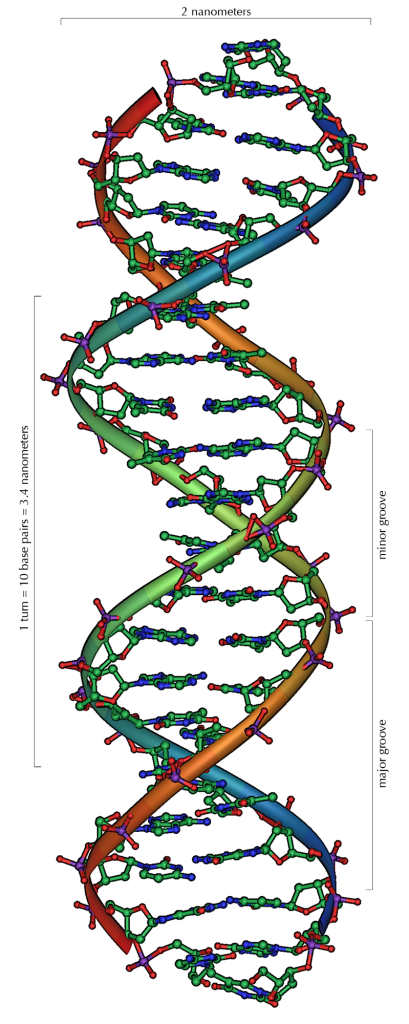
Immagine realizzata da Michael Ströck e rilasciata con licenza libera http://en.wikipedia.org/wiki/File:DNA_Overview.png
Il DNA è un polimero organico costituito da monomeri chiamati nucleotidi. Tutti i nucleotidi sono costituiti da tre componenti fondamentali: un gruppo fosfato, un deossiribosio e una base azotata. Il gruppo fosfato ed il ribosio si legano a formare lo scheletro della doppia elica, mentre i pioli sono costituiti da coppie di basi azotate. Le basi azotate che possono essere utilizzate nella formazione dei nucleotidi da incorporare nella molecola di DNA sono quattro: adenina (A), guanina(G), citosina (C) e timina (T).
L’ordine nella disposizione sequenziale dei nucleotidi costituisce l’informazione genetica.
Possiamo in effetti rappresentare questa informazione come una sequenza di lettere, o stringa di caratteri. Le stringhe di caratteri che otteniamo possono essere analizzate in vari modi e restituire informazioni con un significato biologico.
Il terminale
Versione per i sistemi operativi di tipo Unix (Linux e Mac)
Iniziamo a familiarizzare con questa idea eseguendo dei semplici comandi su un file che contiene le sequenze nucleotidiche di alcuni geni.
Per prima cosa stampate a video il nome della directory in cui vi trovate: il comando pwd (print working directory) vi consente di farlo
Quindi spostatevi nella vostra home con il comando cd ~
Controllate utilizzando di nuovo pwd di avere eseguito il comando con successo e di essere in effetti nella home
A questo punto stampate a video la lista dei files contenuti nella vostra home con il comando ls (list)
Nella home dovreste aver trovato la cartella “esercitazione_bio”, stampate a video la lista dei files presenti in quella cartella digitando il comando ls esercitazione_bio (ricordavi che il TAB è un potentissimo strumento di aiuto!!!)
Ora spostatevi nella cartella dell’esercitazione utilizzando scrivendo cd eser e poi premendo TAB
Iniziamo a scoprire come sono fatti i files contenenti le sequenze di DNA stampandone a video il contenuto: cat sequenze.txt
Se il file è molto lungo e quindi facciamo fatica a leggerlo possiamo iniziare stampando a video solo le prime righe, ad esempio digitando: head -n 20 sequenze.fasta
Un comando simile ci consente di stampare solo le ultime righe del file: tail -n 10 sequenze.fasta
Versione per sistemi operativi di tipo Windows
Iniziamo a familiarizzare con questa idea eseguendo dei semplici comandi su un file che contiene le sequenze nucleotidiche di alcuni geni.
Apriamo il prompt dei comandi nel seguente modo: in un sistema operativo windows 8 o successivo spostate il cursore nell’angolo in alto a destra fino a quando non compare il menu; cliccate sulla lente di ingrandimento “ricerca” e digitate “prompt” ; cliccate su prompt dei comandi e si aprirà la finestra del prompt dei comandi di windows.
In sistemi operativi di tipo Windows delle edizioni precedenti basta cliccare sul pulsate start in basso a sinistra, poi cerca e poi digitare “promt” e selezionare prompt dei comandi.
Per prima cosa osservate il nome della directory in cui vi trovate: il percorso è specificato prima del prompt di comando (il punto in cui si trova e lampeggia il vostro cursore).
Quindi spostatevi nella cartella di lavoro con il comando cd C:\esercitazione_bio
Controllate che il vostro comando sia andato a buon fine e che non abbiate fatto errori. Come? Controllando che ora il percorso che precede il vostro prompt sia quello desiderato: D:\esercitazione_bio> oppure scrivendo chdir e visualizzando la directory dove vi trovate
A questo punto stampate a video la lista dei files contenuti nella cartella operativa con il comando dir
Il tasto tab ( ) è una grande amico, provate a scriverecd C:\eser e poi a spingere il tasto tab .. cosa succede?
) è una grande amico, provate a scriverecd C:\eser e poi a spingere il tasto tab .. cosa succede?
Iniziamo a scoprire come sono fatti i files contenenti le sequenze di DNA stampandone a video il contenuto: type sequenze.txt
Anche in questo caso potreste scrivere type seq e poi a spingere il tasto tab .. Funziona?
Il file contenente le sequenze di DNA
Come avrete notato osservando con attenzione le vostre stampe a video, il file “sequenze” ha la seguente struttura: all’inizio della riga c’è un simbolo di maggiore seguito dal nome della sequenza; poi c’è un accapo; a partire dalla nuova riga c’è una lunga fila di lettere a, c, g, t senza spazi disposta su più righe che rappresenta appunto la sequenza di DNA. Sapreste dire quante sequenze di DNA distinte identificate da un nome ci sono in quel file?
Versione per sistemi operativi di tipo Unix
Con il comando grep “>” sequenze.fasta stampate a video tutte le righe in cui è presente il simbolo “>” (grep è un comando molto utile con le sequenze di DNA, vale la pena approfondire)
E ora per sapere quante sono cosa fate, le contate con il dito dal video? mah no, potete lanciare due comandi in sequenza in questo modo: grep “>” sequenze.fasta | wc -l Per salvare il risultato che avete ottenuto in un file, basta direzionare il risultato del vostro comando in un file grep “>” sequenze.fasta | wc -l > numero_sequenze.txt
Versione per sistemi operativi di tipo Windows
Con il comando find “>” sequenze.fasta stampate a video tutte le righe in cui è presente il simbolo “>” (è un comando molto utile con le sequenze di DNA!)
E ora per sapere quante sono cosa fate, le contate con il dito dal video? mah no, potete lanciare due comandi in sequenza in questo modo: find /c “>” sequenze.txt
Il nostro esercizio di oggi: utilizziamo le sequenze di DNA per capire cosa è successo ai nostri pazienti!
Cosa è successo
Nella città di New York si stanno verificando una serie di casi di tubercolosi. Alcuni malati vengono ricoverati nell’ospedale dove lavora un vostro amico medico, il quale tratta i pazienti con l’antibiotico Rifampicina. Mentre molti dei pazienti mostrano immediatamente segni di miglioramento alcuni non sembrano invece rispondere al farmaco. Per capire la ragione del problema e riuscire a curare i pazienti che peggiorano invece di migliorare il vostro amico contatta il servizio di microbiologia dell’ospedale e gli consegna i campioni prelevati da 5 pazienti che non rispondono all’antibiotico e da 5 pazienti che invece stanno rispondendo alle cure. Nel laboratorio di microbiologia i batteri che infettano ciascuno dei pazienti vengono coltivati e testati per vedere se sopravvivono all’antibiotico. In effetti i batteri che infettano i 5 pazienti che non rispondono alle cure proliferano in coltura nonostante la presenza dell’antibiotico, mentre quelli prelevati dai pazienti in via di miglioramento non sopravvivono in coltura in seguito alla somministrazione dell’antibiotico.
Da 2 colture batteriche (una di un paziente che risponde all’antibiotico e una di un paziente che non risponde) viene estratto il DNA e ne viene determinata la sequenza al fine di indagare meglio questo comportamento.
Le 2 sequenze ottenute vengono inviate proprio a voi, per vedere se da quelle stringhe di caratteri si riescano ad estrapolare informazioni utili. Le sequenze ottenute durante questa indagine si trovano in ciascuna delle vostre home nella cartella esercitazione_bio ed il nome dei file è DNAtubercolosi_sensibile e DNAtubercolosi_resistente (li dovreste aver visti già prima quando avete fatto ls all’interno di questa cartella).
Cosa hanno di diverso le sequenze dei batteri che infettano i pazienti che non rispondono alle cure?
Iniziamo confrontando le due sequenze (DNAtubercolosi_sensibile e DNAtubercolosi_resistente) per vedere se ci sono e quali sono le differenze. Per farlo utilizziamo il programma “Blast 2 sequences”.
Cliccate sul link per accedere al programma e incollate nella due finestre rispettivamente la sequenza del batterio sensibile all’antibiotico e quella del batterio resistente all’antibiotico. Scegliete l’opzione “ e lanciate l’analisi (cliccando sul bottone blu in basso a sinistra “BLAST”)
Ora analizziamo insieme il risultato. Ci sono differenze? Quali? Quali sono i possibili effetti di queste differenze?
Cerchiamo di associare alla nostra sequenza di DNA possibili funzioni
La nostra sequenza di DNA potrebbe contenere geni che codificano per proteine e questi geni potrebbero essere preceduti da promotori. Per verificare la eventuale presenza di geni nei tratti di DNA di nostro interesse, proviamo a tradurre le nostre sequenze in proteine, partendo da tutte le posizioni possibili. Se troviamo una traduzione che ha almeno 50 aminoacidi uno di seguito all’altro senza stop (rappresentati dal programma come asterischi) allora avremo trovato una possibile proteina.
Incollate uno per volta il contenuto dei file DNAtubercolosi_sensibile e DNAtubercolosi_resistente nella finestra di input del programma che si trova a questo link otterrete la traduzione in proteina dei vostri eventuali geni di partenza.
Guardiamo insieme il risultato, ci sono possibili geni? Quale delle possibili traduzioni scegliereste?
(La risposta corretta è: il reading frame 2 on the direct strand. Sapreste spiegare perchè?)
Si tratta di funzioni note?
Se avete trovato dei geni codificanti per proteine durante il passaggio precedente, vale la pena controllare se quelle proteine sono già note o somigliano a proteine note a cui è stata già associata una funzione. Per farlo useremo di nuovo BLAST, il nostro programma per comparare le sequenze fra di loro, ma questa volta con una diversa modalità.
Cliccate su questo link per accedere alla versione del programma BLAST che serve per comparare le sequenze di proteine fra loro e in particolare che consente di comprare la sequenza di una proteina di nostro interesse con le sequenze di tutte le proteine presenti nella bancadati delle sequenze proteiche. Incollate nella finestra la sequenza proteica che avete ottenuto traducendo il DNAtubercolosi_sensibile in aminoacidi scegliendo il risultato ottenuto per il reading frame 2 on the direct strand. Lasciate tutte le impostazioni così come le trovate e cliccate sul bottone “BLAST” infondo a sinsitra.
Guardiamo di nuovo insieme i risultati. La nostra ricerca identifica qualche proteina nota? Qual’è la funzione di questa proteina? Ritenete che sia una funzione importante per la sopravvivenza delle cellule batteriche?
Le differenze che abbiamo trovato fra le sequenze di DNA si traducono in qualche effetto sulle sequenze delle proteine codificate dai geni che abbiamo trovato in quelle brevi sequenze di DNA?
Per rispondere a questa domanda ricorriamo di nuovo all’aiuto di BLAST. Lo useremo di nuovo per confrontare due sequenze fra loro, ma questa volta saranno sequenze di aminoacidi invece che sequenze di nucleotidi.
Cliccate per accedere al programma “Blast 2 sequences” che questa volta è impostato per comparare sequenze di proteine (lo avete notato?) e incollate nelle due finestre rispettivamente la proteina che avete tradotto a partire dal DNAtubercolosi_sensibile e quella che avete tradotto a partire dal DNAtubercolosi_resistente (sempre reading frame 2 on the direct strand).
Ci sono differenze fra le due proteine? Potete ipotizzare qualche interpretazione di questo dato? Perchè un batterio viene ucciso dall’antibiotico e l’altro no?
Cosa potrebbero significare queste differenze?
Con i dati che avete ottenuto proviamo a fare insieme una ipotesi su cosa sta succedendo. Ci ragioniamo insieme
Cosa c’è di vero in questa storia
La resistenza dei batteri agli antibiotici, un problema reale
E’ proprio utilizzando un metodo molto simile a quello che abbiamo descritto in questa esercitazione che si sono scoperte le basi molecolari di alcuni casi comuni di resistenza batterica agli antibiotici. Nel sito che vi ho lincato trovate una tabella che ne elenca alcuni divisi per tipologia.
La resistenza di Mycobacterium tuberculosis agli antibiotici e la nuova ondata di vittime della tubercolosi
Il caso che abbiamo esaminato noi oggi, è un caso reale di resistenza di Mycobacterium tuberculosis alla Rifampicina dovuto ad una mutazione puntiforme (cioè di una singola base del DNA) nel gene che codifica per una proteina fondamentale per la sopravvivenza del batterio: la subunità B della RNA Polimerasi. L’antibiotico Rifampicina si lega a questa proteina e ne blocca l’attività, così i batteri, senza una delle funzioni fondamentali per la sopravvivenza muoiono. Questa singola mutazione che abbiamo visto oggi (e ce ne sono altre note) impedisce alla Rifampicina di legarsi alla subunità B della RNA Polimerasi ma non impedisce alla RNA Polimerasi di svolgere la sua funzione, quindi i batteri continuano a proliferare anche in presenza dell’antibiotico.
Per approfondire sulla tubercolosi e per capire perchè nella mia storia ho scelto proprio New York, potete consultare la pagina di wikipedia dedicata a questa malattia.
La sequenza
La sequenza del gene per la “DNA-directed RNA polymerase subunit beta” di Mycobacterium tuberculosis H37Rv (un ceppo virulento sensibile alla Rifampicina) che avete utilizzato in questa esercitazione è la vera sequenza ottenuta da studi sperimentali e depositata nel database pubblico dell’NCBI. La differenza che avete trovato è una mutazione puntiforme realmente osservata in batteri che non rispondono all’antibiotico. Si tratta di una delle mutazioni sequenziate frequentemente nei soggetti resistenti ed è ampiamente studiata, è nota come mutazione in posizione 531. Il resido in posizione 531 è normalmente una serina, ma nei mutanti viene sostituita da un altro aminoacido. Il residuo 531 corrisponde al 450-esimo residuo amminoacidico della sequenza che vi ho fornito e la vostra mutazione scambia una serina con una prolina.





Pingback: Il progetto Genoma Umano | Sperimentando
Pingback: Bioinformatica per tutti: alla scoperta del mondo dei microbi utilizzando il DNA | Sperimentando
Pingback: Indovina chi – a chi appartiene questo DNA e cosa ci dice di lui | Sperimentando
Pingback: La bioinformatica è uno strumento per l’insegnamento della biologia – Sperimentando
Buon giorno,
vorrei cimentarmi in questa esercitazione ma non capisco dove trovare i file di partenza citati per creare la cartella esercitazione_bio. Può darmi indicazioni per favore? Complimenti per il bellissimo lavoro.