Le sequenze di DNA
Nell’unità di apprendimento precedente “le sequenze di DNA sono stringhe di caratteri” abbiamo imparato che:
- la sequenza del DNA di una cellula è necessaria e sufficiente per determina come funziona e come appare quella cellula. Cosa succede infatti se sintetizziamo il DNA di un certo organismo e lo infiliamo nel nucleo di una cellula privata del suo DNA? La seconda cellula a poco a poco diventa uguale all’organismo da cui abbiamo copiato il DNA, lo ha fatto Craig Venter e a questo link potete leggere la notizia riportata da Scientific American! (“uguale all’organismo da cui abbiamo copiato il DNA” questa affermazione è vera QUASI del tutto.. – video sulle ultime frontiere dello studio del DNA: l’epigenetica)
- la sequenza del DNA può essere rappresentata come una sequenza di lettere, ovvero un testo, che possiamo studiare e analizzare con l’aiuto dell’informatica.

Riprendiamo proprio dove ci eravamo lasciati facendo un gioco: indovina chi

Vi fornirò una sequenza di DNA, sarà come avere una carta coperta che rappresenta la persona o l’animale o il microbo che dovete indovinare. Facendo le giuste domande riuscirete ad indovinare che apparenza ha e come è fatto l’individuo nascosto. Immagino che abbiate già giocato ad indovina chi no? questa sarà la versione “zoo”, non è detto che chi si nasconde dietro la carta sia necessariamente un individuo del genere Homo, potrebbe appartenere a qualsiasi specie!

Eccovi la carta incognita da mettere in testa: sequenzaDNAignota.fasta
Ecco come facciamo passo per passo a indovinare di chi si tratta:
- apriamo il file con la sequenza e leggiamo l’intestazione (vi ricordate l’intestazione?)
- copiamo la sequenza e usiamo un algoritmo di allineamento chiamato “Blast” per confrontare la sequenza con la banca dati di sequenze note e raccogliere informazioni:
- cliccate su questo link per accedere al programma blast on line
- incollate la vostra sequenza nella finestra in alto”Enter accession number(s), gi(s), or FASTA sequence(s)”
- quindi scegliete di cercare nella banca dati che contiene tutte le sequenze di nucloetidi (è l’impostazione standard, non occorre modificare niente)
- cliccate infondo a sinistra sul bottone “BLAST” e attendete il risultato
- ora leggiamo insieme il risultato del blast e cerchiamo di rispondere a queste domande:
- c’è una sequenza identica alla nostra nella banca dati?
- che funzione codifica il nostro gene?
- qual’è il nome scientifico dell’organismo a cui appartiene?
- questo organismo ha i baffi? e gli occhiali? è pelato?
- qual è il suo nome comune?
- Scrivete le risposte a queste domande in un file blocconote
Se il gioco vi è piaciuto potete ripeterlo con questa nuova carta, le procedure sono le stesse e queste saranno le domande:
- c’è una sequenza identica alla nostra nella banca dati?
- che funzione codifica il nostro gene?
- a che classe di organismi appartiene il nostro organismo ignoto?
- sapreste trovare qualche immagine degli organismi di questa classe in rete?
- riportate la risposta nel solito file blocconote che avete aperto prima
Ora cambiamo un po’ il gioco e andiamo in cerca di una persona. Sì, avete capito bene, di una persona con nome e cognome a partire da qualche sequenza di lettere del suo DNA. Questa è la SequenzaUmanaIgnota e ora vi spiego come facciamo:
- apriamo il file con la sequenza in un nuovo tab
- copiamo la sequenza e usiamo “Blast” per confrontare la sequenza con la banca dati di sequenze note, ma stavolta scegliamo accuratamente la sezione della banca dati che ci interessa:
- cliccate su questo link per accedere al programma blast on line
- incollate la vostra sequenza nella finestra in alto “Enter accession number(s), gi(s), or FASTA sequence(s)”
- quindi scegliete di cercare nella banca dati “Whole-genome shotgun contigs (wgs)” usando il menù a tendina. Si tratta della raccolta dei pezzettini di sequenze ottenute durante i progetti di sequenziamento di interi genomi
- inoltre decidete di limitare la ricerca alle sole sequenze umane, visto che possedete già questa informazione. Potete farlo cliccando sull’opzione “organism” accanto alle parole “limit by” e scrivendo nella finestrella “Homo sapiens (taxid:9606)”
- cliccate infondo a sinistra sul bottone “BLAST” e attendete il risultato
- ora leggiamo insieme il risultato del blast e cerchiamo di rispondere a queste domande:
- c’è una sequenza identica alla nostra nella banca dati?
- cliccando su questa sequenza e seguendo i link otteniamo una pagina ricca di informazioni.. che ci serviranno per rispondere alle seguenti domande:
- la persona in questione ha i capelli o è pelata?
- ha i baffi?
- che lavoro fa?
- perchè lo conosciamo e qual’è il suo nome?
Dal 2008 ad oggi, i genomi di molti individui sono stati sequenziati. Molti di questi dati sono privati, ma molti altri sono a disposizione nelle banche dati pubbliche. Le ragioni principali per cui sequenziamo riguardano:
- il nostro interesse per le peculiarità delle persone provenienti da aree geografiche diverse. Questo ad esempio ci ha portato a scoprire che le razze umane non esistono e molto altro
- il nostro interesse per le malattie che ancora non sappiamo spiegare e curare. In tutto il mondo da anni si stanno costruendo mappe della localizzazione sul genoma di nucleotidi associati a stati patologici, magari in combinazione con altri. Tutti questi dati sono pubblici e costanetemente aggiornati.. ma il completamento della mappa è ancora lontano..
E voi, che informazioni cerchereste nei genomi?
“L’impronta digitale”
Ognuno di noi ha un aspetto e un funzionamento molto complessi, ma abbiamo cercato un modo per identificare una persona senza doverla guardare proprio tutta, guardando solo un piccolo dettaglio che è unico per ciascuno di noi e pertanto ci identifica. Ad esempio l’impronta delle nostre dita sembra essere un dettaglio abbastanza unico, pertanto prendendo l’impronta digitale ad uno sconosciuto e confrontandola con le impronte presenti in una banca dati, possiamo dire se quell’individuo corrisponde ad una delle persone schedate ed eventualmente chi è, senza bisogno di prendere le misure di tutti i dettagli della sua intera persona.
Nello stesso modo ci siamo chiesti: è possibile leggere un solo pezzetto di DNA senza doverlo leggere proprio tutto, per poter dire, difronte ad un organismo sconosciuto, se lo abbiamo già incontrato e di chi si tratta?
La risposta è che è possibile e abbiamo trovato vari modi per farlo con gli individui della specie umana. Una delle tecniche più diffuse si chiama DNA fingerprinting
Ma proviamo a fare un passo indietro e proviamo a pensare insieme a che caratteristiche dovrebbe avere questo segmento di DNA se volessimo ad esempio identificare la specie.
Dovrà essere:
- presente in tutti gli organismi
- uguale fra individui della stessa specie
- diverso fra organismi di specie diverse
La grande scoperta di Carl Woese!
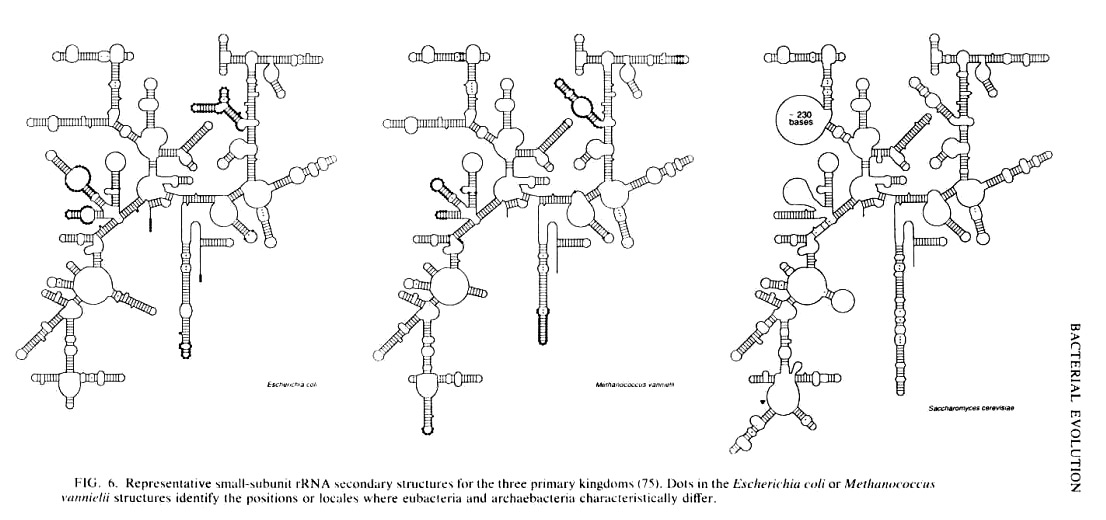
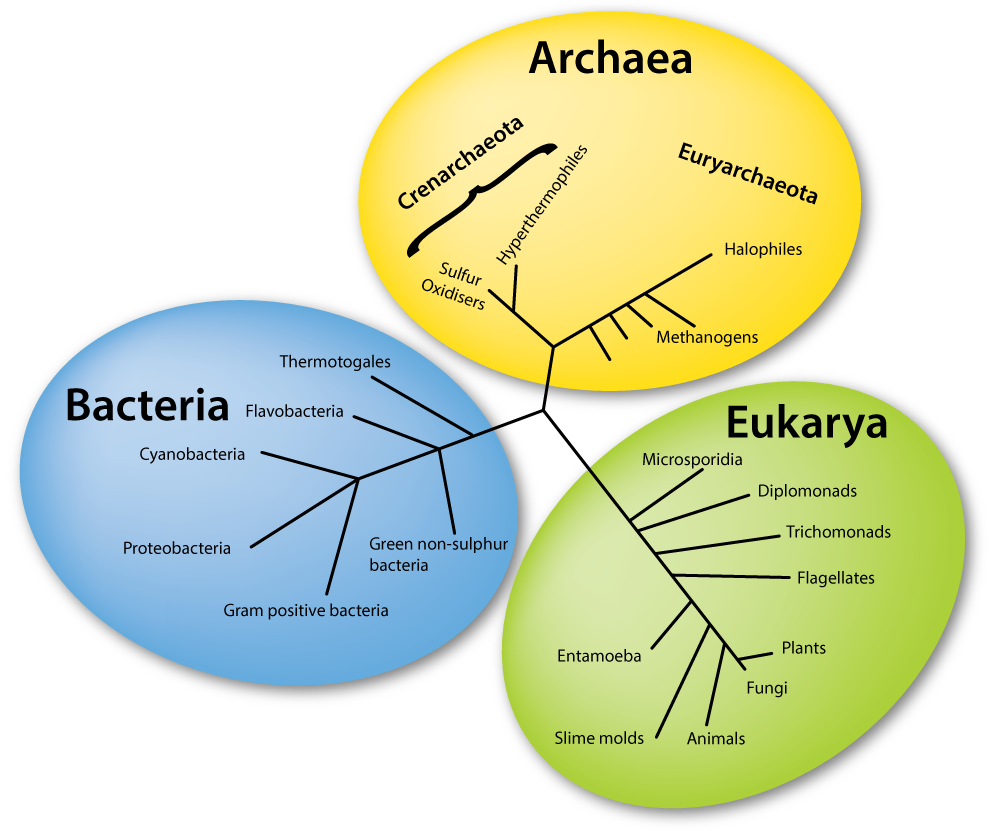
Ora proviamo noi a sperimentare questo sistema di identificazione e a guardare le parentele
“Mi dia la sua impronta digitale se vuole superare il check-in”
Ci troviamo nell’ufficio controlli di un aeroporto e dobbiamo controllare il carico di un volo intercontinentale. Durante il periodo Natalizio un laboratorio di Padova spedisce ai colleghi di Sidney alcune piastre piene di microrganismi colorati.

Non vogliamo assolutamente che specie di batteri infettivi e pericolosi per la salute vengano spostati da un continente all’altro, pertanto analizziamo la sequenze dei geni per gli RNA Ribosomali 16S di ciascuno degli organismi presenti nelle piastre prima di autorizzare l’imbarco.
Questo è il file con le sequenze che il nostro laboratorio di controlli ha prodotto. Incollate nella pagina di Blast una per una le singole sequenze, scoprite se corrispondono a organismi noti e quindi determinate, aiutandovi con una ricerca nella rete, se si tratta di organismi patogeni o infettivi o comunque pericolosi per la salute dell’uomo.
Qual’è la lista degli organismi strisciati sulle piastre?
Che cosa “fanno” questi organismi?
Secondo le vostre analisi, possiamo imbarcare la spedizione?





Pingback: microbi, comunità microbiche e metagenomica | Sperimentando
Pingback: La bioinformatica è uno strumento per l’insegnamento della biologia – Sperimentando