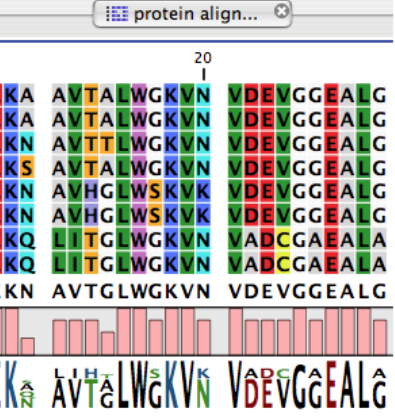
 La bioinformatica si fonda su un’idea molto semplice ma molto potente: le macromolecole biologiche sono polimeri costituiti da sequenze di monomeri concatenate far loro, se assegno ad ogni monomero una lettera ogni polimero è una parola. Le parole in quanto tali possono essere analizzate con tutti gli strumenti informatici che abbiamo a disposizione per analizzare files di testo.
La bioinformatica si fonda su un’idea molto semplice ma molto potente: le macromolecole biologiche sono polimeri costituiti da sequenze di monomeri concatenate far loro, se assegno ad ogni monomero una lettera ogni polimero è una parola. Le parole in quanto tali possono essere analizzate con tutti gli strumenti informatici che abbiamo a disposizione per analizzare files di testo.
Una delle cose che facciamo più frequentemente è confrontare le parole fra loro per vedere se si somigliano, quanto si somigliano, se ci sono gruppi di lettere in comune fra parole associate ad una stessa funzione o ad una stessa localizzazione cellulare.
Ad esempio, c’è qualcosa di ricorrente nelle sequenze che precedono il sito di inizio della trascrizione? Se ci fosse avremmo trovato la firma delle regioni promotoriali e potremmo prevederle in un nuovo tratto di DNA che sequenziamo e di cui non sappiamo ancora niente.
Il sequenziamento di molecole biologiche, in particolare DNA ed RNA, ma anche proteine è diventato sempre più veloce e più economico negli ultimi anni. Proprio per questo è diventato il metodo prediletto per tante indagini di tipo biologico. Le applicazioni di questa tecnica sono vastissime e vanno dalle ricerche “discovery driven”, sequenziamo e vediamo se i dati che raccogliamo ci fanno vedere cose che neanche immaginavamo (ad esempio il sequenziamento del genoma umano ha aperto molte più nuove domande di quante ne abbia risposte) alle ricerche “hypothesis driven”, usiamo il sequenziamento all’interno di un esperimento per trovare risposte a domande che ci poniamo da anni. Il numero di applicazioni che attualmente sono state immaginate e sono in uso è molto vasto, ma non c’è limite alla fantasia, soprattutto a quella degli studenti! Per cui il suggerimento è raccontate loro che esiste il sequenziamento e spiegate con esempi quali informazioni riusciamo ad estrarre dai dati di sequenziamento. Infine invitate loro a immaginare esperimenti in cui userebbero questa tecnica.. non rimarrete delusi!
Perchè introdurre la bioinformatica nelle scuole superiori
Contenuti che gli studenti possono acquisire
- la bioinformatica si presta molto bene per affrontare molti concetti di biologia molecolare, biologia umana, genetica ed evoluzione
- iniziare ad usare le banche dati e molti software di analisi bioinformatica dà agli studenti un insieme di strumenti che saranno utilissimi durante gli studi universitari scientifici e che gli saranno utili per analizzare le notizie di attualità
- I contenuti sono diversi nei vari curriculum di studi:
-
- insegnare a sviluppare programmi per analizzare dati (riguarda una collaborazione fra insegnanti di scienze e di informatica)
- insegnare ad utilizzare programmi preesistenti per analizzare un particolare gruppo di dati (può essere completamente in carico dell’insegnante di scienze)
Utilità metodologiche
- la bioinformatica è un ottimo metodo per far esercitare gli studenti con il metodo sperimentale
- si può applicare a domande molto concrete e attuali
- utilizza strumenti moderni che gli studenti conoscono bene
- molte applicazioni sono estremamente interattive
- si presta per assegnare agli studenti percorsi differenziati e compiti individuali
Ambiti di studio in cui si può usare questo metodo
- Filogenesi
- Geni, RNA, caratteristiche dei trascritti
- Genetica umana: varianti geniche e geni malattia
- Enzimi e proteine
- Mappe del metabolismo cellulare
- scoperta e analisi di comunità microbiche
Come usare questo strumento:
UN PO’ DI CONSIGLI
La bioinformatica non è una materia per se, non è l’ennesimo tassello da aggiungere ad un programma già molto denso, ma è uno strumento nelle mani dell’insegnante per affrontare con gli studenti in modo interattivo gli argomenti di lezione. Soprattutto è uno strumento importante per fare scienza in classe, per non raccontare l’argomento, ma scoprire i contenuti insieme agli studenti grazie ad esperimenti già fatti o da fare a portata di click!
Quindi non ci sono attività già pronte, ma ci son attività che l’insegnante può preparare di volta in volta, che può disegnare seguendo 10 regole d’oro che David Form and Fran Lewitter ci indicano in un articolo pubblicato nel 2011 su Plos Computational Biology intitolato “Ten Simple Rules for Teaching Bioinformatics at the High School Level”. (Una versione in italiano di queste 10 regole d’oro è disponibile fra le diapositive del corso per docenti tenuto ad Orvieto (TR), a San Bonifacio (VR) e a Perugia)
UN PO’ DI ESERCIZI PER IMPRATICHIRSI
Dobbiamo imparare a padroneggiare alcuni strumenti per potere preparare delle esercitazioni interessanti per gli studenti.
Il primo obiettivo è certamente imparare a cercare informazioni nelle banche dati di interesse biologico. Provate a fare qualche ricerca nelle seguente banche dati:
NCBI (http:// www.ncbi.nlm.nih.gov/)
EBI (http://www.ebi.ac.uk/)
PDB (http://www.rcsb.org/ pdb/home/home.do)
Paleobiology Database (http:// fossilworks.org/?a=home)
PubMed (http://www.ncbi.nlm.nih.gov/pubmed)
QUALCHE LEZIONE GIA’ PRONTA
Infine in effetti non ha senso ripreparare tutti mille volte la stessa lezione. Per cui metto a disposizione di tutti gli insegnanti di scienze le lezioni pubblicate su questo sito.
In particolare c’è una lezione che riguarda gli RNA che non codificano per proteine, nella quali si parte dalle evidenze in banca dati per scoprire quali RNA esistono e poi discutere quale sia la funzione.
Una ottima esercitazione introduttiva alla bioinformatica per docenti e studenti della secondaria di secondo grado è questa: mutazioni e resistenza agli antibiotici per studenti senza prerequisiti di informatica. In questa esperienza viene presentato un file di sequenze di DNA, si apprendono semplici passaggi per analizzarlo utilizzando un computer e si iniziano ad usare alcuni software disponibili online per confrontare sequenze di DNA, identificarle, attribuirgli una funzione, tradurle in proteina. Alla fine dell’avventura si riuscirà a caratterizzare una forma di resistenza all’antibiotico e si cercheranno soluzioni esistenti o futuribili per curare il paziente. L’esercitazione richiede 2 ore.
Una variante di questa esperienza pensata per studenti con conoscenze di programmazione utilizza in parte software accessibili online e in parte programmini scritti dagli studenti insieme all’insegnante di informatica. L’esercitazione in questo caso richiede circa 4 ore in tutto.
L’introduzione alla bioinformatica nel caso degli studenti della scuola secondaria di primo grado può essere fatta con questa esperienza dedicata alla biodiversità che utilizza software disponibili online per comparare e identificare sequenze di DNA e per localizzare sulla carta della Terra il ritrovamento e la datazione di fossili delle varie specie.
Per parlare di biodiversità e biodiversità microbica nella scuola secondaria di secondo grado ci sono queste esercitazioni da due ore ciascuna: a chi appartiene questo DNA; paleontologia interattiva; microbi comunità microbiche e metagenomica.
Per parlare delle migrazioni dell’uomo, degli elementi trasponibili e dell’evoluzione dei genomi l’analisi delle sequenze Alu è sicuramente un ottima esercitazione, che può essere fatta solo al computer o anche in laboratorio.
Per rispondere ad una richiesta dei miei studenti riguardo ad alcuni fatti di attualità che li avevano colpiti ho preparato una lezione sulla ricerca delle informazioni su internet. A questa lezione è anche seguita una lezione sul biorisanamento in cui sono stati chiamati loro a mettere delle informazioni su internet.
Nella lezione riguardo al genoma umano abbiamo utilizzato molti strumenti messi a disposizione dal consorzio internazionale genoma umano che prevedono una speciale sezione riguardo alla bioinformatica (questo materiale è tutto in inglese).
Spero che questo sito si arricchisca presto di nuove esperienze, se preparate voi delle altre lezioni, condividetele pure su questo sito, sarà lavoro prezioso per tutti #bioinformatica!
Infine cliccando su questo link troverete le diapositive del corso