Sequenziatori: i nuovi microscopi
Leggere il DNA, abbiamo visto, ci dà molte informazioni:
- dice tutto quello che fa una cellula
- dice come appare quella cellula
- dice chi è l’organismo, lo identifica
Se vogliamo conoscere i segreti di un organismo non serve quindi spezzettarlo e guardare ogni suo frammento sotto la lente di un microscopio, ci basta un po’ del suo DNA e possiamo leggere la sequenza per raccogliere molte informazioni!
Inoltre se siamo in luogo e vogliamo sapere chi c’è, beh gli organismi grandi li vediamo con i nostri occhi, ma quelli piccoli? o ci portiamo un microscopio, oppure raccogliamo un campione e leggiamo tutto il DNA che c’è dentro. Potremo usare l’RNA ribosomale come “impronta digitale” per fare l’elenco dei presenti, oppure leggere tutto e scoprire che cosa sono capaci di fare quei microrganismi presenti, che programma hanno!
Vi viene in mente qualche luogo dove andreste a rovistare?
Beh agli scienziati ne sono venuti in mente davvero tanti, ora ne vedremo insieme qualcuno e che cosa hanno scoperto!
Applicazioni: alla scoperta di un mondo microscopico invisibile o poco visibile ai nostri occhi
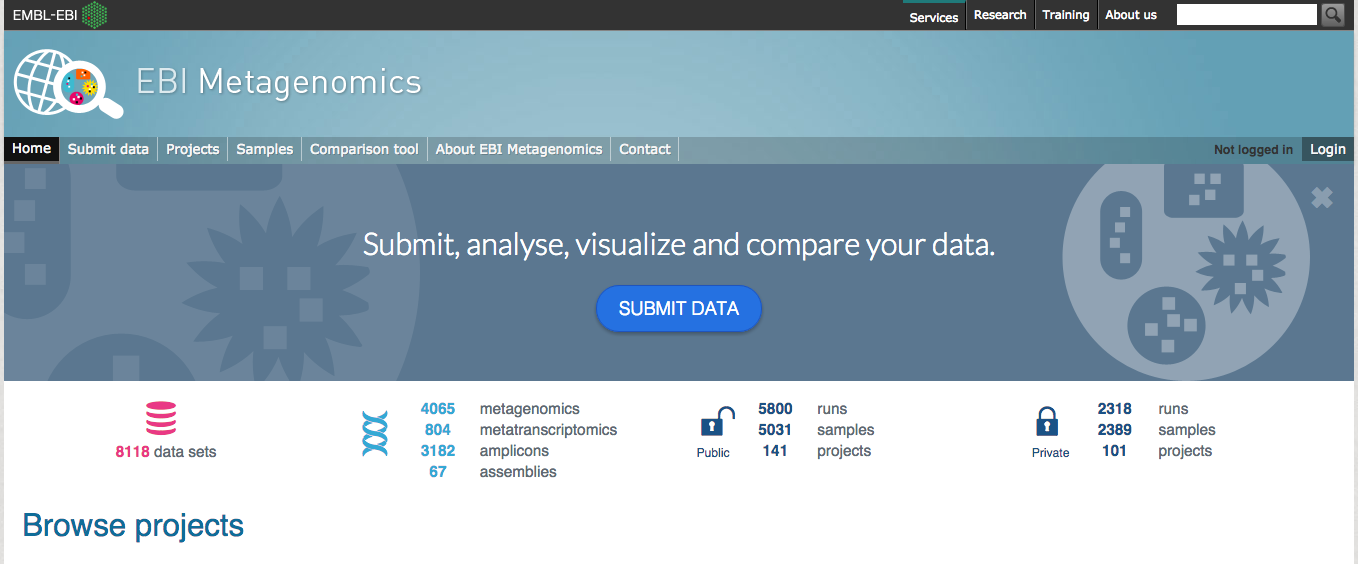
Ora facciamo noi qualche studio da bioinformatici. Sul portale “metagenomics” dell’Istituto Europeo di Bioinformatica sono accessibili i dati originali di molti diversi studi e sono anche disponibili dei programmi di analisi on line.
Diamo un’occhiata insieme:
Cosa succede ai nostri inquilini intestinali quando prendiamo gli antibiotici?
Come abbiamo appena visto le superfici interne ed esterne del nostro corpo sono popolate da microrganismi che contribuiscono alle nostre funzionalità corporee. In qualche caso però anche i batteri patogeni, che normalmente non risiedono nel nostro corpo, si uniscono alla festa e iniziamo a star male. Il modo migliore che abbiamo per curarci è assumere delle sostanze cha hanno un effetto nocivo sulle cellule batteriche ma nessuno o trascurabili effetti sulle nostre. Perfetto per noi, ma cosa ne è dei nostri ospiti abituali, i batteri dell’intestino e della pelle?
Alcuni scienziati hanno raccolto campioni dal tratto digerente di individui sani che non assumevano antibiotici e quindi li hanno raccolti di nuovo diversi giorni dopo l’assunzione di un particolare tipo di antibiotici. I dati sono disponibili nel portale che abbiamo appena visto insieme.
Al centro della pagina a sinistra c’è un breve elenco dei progetti divisi per categoria, cliccate su “Human digestive system”
apparirà una lista degli effettivi progetti riguardanti il tratto digerente per cui sono disponibili i dati, scegliete “Beta Lactam Antibiotics and Human Gut Microbiota”
La pagina del progetto a cui avete appena avuto accesso offre una breve descrizione dell’esperimento e delle principali conclusioni, subito sotto troviamo la tabella dei dati sperimentali. Proviamo a guardare insieme alcuni dati:
- nella riga “Human gut metagenome, 0 days after antibiotics” guardiamo prima la pagina corrispondente ai dati sperimentali
- sempre nella riga “Human gut metagenome, 0 days after antibiotics” guardiamo ora la sezione “Taxonomy” del secondo dei due esperimenti “Metagenomic”
- dopo aver esaminato in dettaglio questi dati andiamo ora a guardare la stessa sezione “Taxonomy” del secondo esperimento “Metagenomics” fatto 11 giorni dopo la somministrazione degli antibiotici
Sapreste dirmi se notate differenze e se si quali?
- guardate ora la sezione “Taxonomy” di uno degli altri giorni a scelta
Confrontatevi fra di voi raccontandovi e discutendo le vostre osservazioni
Per gli stessi due esperimenti ora proviamo a confrontare i dati ottenuti guardando gli interi genomi della comunità microbica con i soli geni per gli RNA ribosomali della stessa comunità.
- confrontate i metagenomi del giorno 0 con gli ampliconi del giorno 0 ; i metagenomi del giorno 11 con gli ampliconi del giorno 11
- riformulate anche il confronto fra giorno 0 e giorno 11 alla luce di entrambi i dati e parliamone insieme
Ora è tempo di utilizzare un nuovo strumento: proviamo a comparare nel nostro esperimento quali funzioni vengono attivamente espresse in varie condizioni:
- nella barra in alto delle pagine del portale che state utilizzando, una delle voci è “Comparison Tool“, cliccate per accedere a questa sezione
- ora selezionate gli esperimenti da comparare. Scegliete dal menu a sinistra il nome del progetto”Beta Lactam Antibiotics and Human Gut Microbiota”
- Nella finestra a destra potete ora scegliere gli esperimenti “metatranscriptome” fatti al tempo zero e i due fatti dopo 11 giorni dalla somministrazione dall’antibiotico
- cliccate in basso al centro il bottone blu con scritto “compare”
Osservate delle differenze? Quali?
Ora se volete potete aggiungere alla comparazione anche i dati del giorno 6 e del giorno 40. Proviamo a commentare insieme quello che vediamo:
- osservazioni
- Potete mettere i risultati di questa comparazione in relazione con i risultati delle comparazioni che abbiamo fatto in precedenza, quando abbiamo guardato quali batteri erano presenti al tempo 0 e dopo 11 giorni?
- perchè abbiamo scelto di comparare i metatrascrittomi piuttosto che i metagenomi ? Se comparate i metagenomi cosa osservate?
Mondo virtuale o mondo reale?
Se pensate che quello che abbiamo fatto in questa sezione sia un gioco, voglio raccontarvi una storia. In alcuni casi in ospedale le persone particolarmente debilitate presentano infezioni batteriche che possono solo essere sconfitte con tanti diversi antibiotici, con una cura ad ampio spettro. In questi casi la gran parte dei nostri abituali inquilini intestinali muore, lasciando così crescere e proliferare pochi batteri resistenti agli antibiotici. L’unico modo che conosciamo al momento per ripristinare una generale condizione di salute e quello di far ricrescere i nostri inquilini, e uno dei modi beh.. è effettuare un trapianto di feci da una persona sana, che serva come inoculo!
Suoli, un mistero ancora in gran parte da svelare
Nella raccolta dei dati sperimentali sui microbiomi è presente un esperimento che compara suoli acidi e suoli basici. Come forse avrete sentito su questi tipi di suoli crescono colture molto diverse..
Dopo aver letto l’overview selezionate “Analysis summary”
Cliccate su Phylum level taxonomies (TSV) e anche su Taxonomic assignments (TSV) per scaricare i dati sul vostro computer.
Ora aprite i files come fogli di calcolo (ad esempio utilizzando Excel) iniziando con i dati Phylum level taxonomies (TSV)
Colorate tutti i campioni dei suoli acidi con un colore e di quelli basici un’altro oppure rinominatele. Utilizzando dei semplici descrittori statistici oppure dei grafici cercate di capire se ci sono differenze significative fra le due categorie di campioni.
Aperta la discussione:
- abbiamo in questo esperimento evidenze di differenze rilevanti ? Se si, quali?
- nei suoli acidi e nei suoli basici vivono gli stessi organismi?
ripetete le analisi sul file Taxonomic assignments (TSV) e cerchiamo di approfondire le osservazioni che abbiamo già discusso
Perchè in questo caso non facciamo il confronto fra le funzioni principalmente rappresentate nei due campioni utilizzando lo strumento “compare”?
Vita in Ambienti Estremi come non la avete mai vista prima!
Fino a qualche anno fa pensavamo che nei deserti salini non ci fosse vita. Molti dei batteri che conosciamo muoiono non appena vengono trasferiti in un ambiente simile. Quando abbiamo iniziato a guardare a questo ambiente con la nuova lente di ingrandimento della metagenomica abbiamo scoperto che il deserto non èera poi così deserto come sembrava.. provate a dare un’occhiata voi stessi cercando sul portale parole chiave come “desert” o “hot spring” o “volcano” o “antarctic” .. o qualsiasi luogo vi sembri estremo.
Possiamo essere tutti coinvolti in un grande esperimento per capire sempre di più la vita sul nostro pianeta
Ogni anno durante il solstizio d’estate i ricercatori di molte diverse località nel mondo raccolgono campioni dall’oceano. Misurano molti parametri chimici e fisici e poi fanno un’analisi di tutti i microrganismi presenti nel campione studiando proprio tutto il DNA che riescono ad estrarre. Si chiama ocean sampling day. A questo progetto è associata una iniziativa di “citizen science” che si chiama MyOSD: con un kit fa da te e una app sul cellulare, ciascuno di noi può contribuire con dati utili a fare una fotografia del pianeta in quella giornata e a fornire la base per nuove ed entusiasmanti scoperte!




Pingback: paleontologia interattiva | Sperimentando
Pingback: La bioinformatica è uno strumento per l’insegnamento della biologia – Sperimentando